海归学者发起的公益学术平台
分享信息,整合资源
交流学术,偶尔风月

“合成数据”或“数据制造”似乎是化学领域的“新概念”。对于科学家来说,伪造或编造数据有着明显的含义,并且这些含义通常并不好。例如,某著名期刊撤回了Sawamura团队2020年的一篇文章,另一著名期刊撤回了Sawamura团队2022年的两篇文章[1];哥伦比亚大学Dalibor Sames教授因其研究生Bengu Sezen造假、编造和抄袭数据导致六篇文章被撤回[2]等等。然而,与造假的编造数据不同,“合成数据”是出于合理的理由,系统性创建的数据。

图源:© JASON FORD @ HEART AGENCY
合成数据对于大多数化学领域来说是不熟悉的词汇,特别是“合成”在化学中有着特定的含义。但事实上,所有的实验研究都会通过设计实验、改变实验条件等来制造产生数据的方式。另一方面,合成数据对于计算化学家来说则很熟悉。计算化学家和所有领域中的计算科学家一样,通过模型和理论以及在计算机上实现的计算,产生数据,来解释和预测各种现象。因此,泛泛地讲,计算科学家的工作首先是制造数据、合成数据。同时,近年来,合成数据对于机器学习和人工智能越来越重要。如大语言模型正在耗尽数据,为了训练下一代模型,需要更大、更新的人类语言和知识数据集,即使是互联网和文学作品中的数十亿词汇也不足以满足这些模型日益膨胀的胃口。解决方案之一便是通过机器模型生成数据集来训练其他机器,构建“无限的数据生成引擎”,合成数据,让这些贪婪的模型自行进食。
在医学和金融等领域,合成数据通常被用来代替来自真实人物的真实数据,以解决隐私问题,或处理数据不平衡问题。例如,当某些族裔的人群在数据集中的代表不足,个人医疗记录等数据量不足或详细程度不够时,通过合成数据以及模型的训练来反映真实数据的统计特征。合成数据从真实数据中来,但不回溯到原始的个人数据。与之相对的,在化学问题中,合成数据更多地与分子的行为有关而不是人有关,因此,合成数据的使用基于不同的原因。“合法编造”数据在化学领域并不是新鲜事,分子设计以及“模拟”或“计算”长期以来一直在产生数据以扩展对化学空间的探索。现在不同的是,无论是合成、模拟还是计算“制造”的数据都广泛被用来与机器学习模型结合使用。AI 模型是能够理解大量数据的算法,而所需的训练集可以是真实的实验数据或者制造出来的数据,或者是两者的综合。AI模型学习数据中的各种模式,并使用它们进行分类和预测,从而提供有价值的新的数据和见解,无论机器是如何得出这些见解的。因此,合成数据不但是 AI 模型的输入,也是 AI 模型的输出。Jacobsen将合成数据定义为“由算法生成,也用于算法”的数据[3],虽然化学家可能并不关心如此严格的定义。例如,一些合成数据的创建技术与“深度伪造”有关,像机器生成逼真的形象和语音一样,生成化学结构。Antunes,Butle和Grau-Crespo开发的APP利用大语言模型,通过对话提示,输入化学式,由机器产生化合物的假想晶体结构[4]。这类应用的更高级形式是预测具有目标性质的化合物、材料和生产可行性。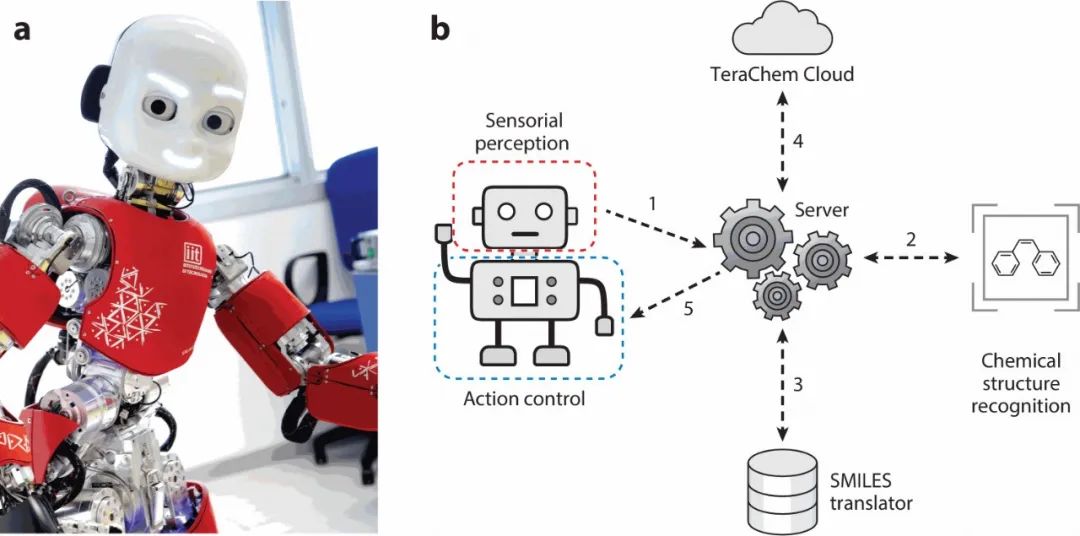
图源:Annu. Rev. Phys. Chem.74:313-36
SMILEStranslator
合成数据的另一个例子是斯坦福大学Martinez团队开发的ChemPix[5],通过识别手绘化学结构来给出机器可读的分子的SMILES表达式。机器学习需要的是大量数据,而真实手绘化学结构所能构建的数据集是有限的。Martinez等人采取的策略是,通过RDKit产生50万个化学结构图像,将其通过图像增强、图像降质以及背景添加等一系列图像处理,制造了合成数据。将合成数据与真实手绘合并,训练机器学习的结果实现了对手绘化学结构识别的70%的准确率。而单纯用干净的RDKit图像训练仅能实现56%的准确率。Martinez等进而在综述文章[6]中探讨了机器学习、图像处理以及云计算支持的交互式量子化学计算。正如Martinez所说,“化学领域中有很多问题——实际上,我认为大多数问题——缺乏足够的数据来真正应用机器学习方法。”,使用合成数据可以节省的成本可能比分子识别示例中的成本要高得多,因为在其他地方,这些制造的数据不仅仅是用来替换那些花费几秒钟才能绘制的数据,而是来自昂贵的真实实验中的数据。
计算机可以为未知的无机化合物(如KRb)生成看似合理的结构 KRb2TiF6(左)和 LiTa2NiSe5(右)
图源:© Luis M Antunes et al
另一方面,即使有海量的实验数据,如果未经标签化,对机器学习来说也毫无意义,因为需要训练机器学习模型识别标签,从而产生有价值的新的数据。例如,英国卢瑟福阿普尔顿实验室历年来产生了千兆级的中子散射数据,但对这些实验数据的标签化非常昂贵,而合成数据则可以同时对数据产生标签。在解释真实光谱方面,理论计算的干净结果同样不适于机器学习,也需要像Martinez团队制作假的手绘化学结构一样,对理论计算结果进行粗糙化。Butler等人[7]将包括噪声在内的实验伪影添加到干净的理论计算数据中,通过由此得到的合成数据,对深度神经网络做了训练,首次实现了对非弹性中子散射实验数据的机器学习分析。他们的工作表明了可解释的机器学习可以是分析非弹性中子散射的有力工具。合成数据与AI的结合可能产生重大影响的另一个领域是药物发现。如在Gurvic, Leach和Zachariae的研究中[8],首先通过已知抗生素训练了一个机器学习模型,有效地预测了针对革兰氏阴性菌的其它化合物;进而将这些预测的化合物作为合成数据,作为输入数据,通过机器学习来预测有药物活性的分子结构单元和化学特征,包括胺、噻吩和卤素等基团。而这些对于寻找新的革兰氏阴性菌抗生素意义重大。合成数据的直观目的是为机器学习模型提供大数据集。合成数据的“隐藏”动机是通过足够的输入数据让我们能够理解为什么机器学习模型做出了它们做出的预测,即机器学习模型的可解释性。AI 模型的内部运作被广泛称为黑盒子,但合成数据可以做为一种工具,来锐化特定的模型,或者以更简单的形式理解其本质。Martinez,Zachariae和Butler的工作都体现了这一点。但并非所有的类似工作都明确地表达了这一目的或者动机。在机器学习领域使用非真实数据的风险现在仍难以确定。在其他领域,合成数据的风险通常与谁产生数据有关,或者与谁受到合成数据产生的预测的影响从而做出决策有关。正如Jacobsen所说,合成数据的风险因应用领域而异,“化学家必须严格界定‘我们如何在特定的语境中界定风险’”。而Zachariae则认为,从纯粹的科学角度来说,合成数据与机器学习之前的通过物理模型实现的预测循环没有什么不同;当然任何预测的目标药物分子都必须经过严格的安全检验。
手绘数十万个化学结构太耗时了,为什么不让计算机来做呢?
图源:© 2021 Hayley Weir et al
合成数据的风险:如果预测与现实不符,将会失去可信度在理论化学领域,实际上没有“真实的”或实验数据可以用来训练机器学习模型,数据集只能通过计算方法来获得,如DeepMind发展的DM21密度泛函[9]。或者,通过小分子的量子化学计算,训练机器学习模型以更快的速度和更低的成本预测大分子的结构和能量。但现有的理论方法对小分子的计算精确度优于对大分子的计算,在缺乏实验验证的情况下,通过理论计算数据集训练的机器学习模型预测的大分子及其能量有可能并不好,进一步说,如果由此机器学习模型预测了上百万的数据点,就可能是个问题。Martinez认为,计算量子力学领域中的合成数据训练集通常会有严格的精度控制,而这可能会给数据正确性和覆盖范围带来错误的信心。在DeepMind 的另一研究中[10],基于过去ICSD,Materials Project以及OQMD多年连续的研究确定的48,000种稳定晶体结构及其中已经实验验证的736种结构,通过机器学习产生了两百二十万种新的层状材料和固体电解质材料。但如果现有的理论并不一定那么准确,如果预测材料的性质与现实不符,将会失去信誉和资金。因此,虽然“编造”数据在现在可能意味着不同的事情,但如果做得不好,仍然存在很大风险。Glorius和Grzybowski等人近期的文章[11]对逆合成的AI做了综述,他们强调,文献中缺乏负面结果和反应的多样性,是对有用的逆合成AI算法的关键障碍。这些模型只与其所用数据集一样好,而那些依赖实验记录的模型尤其容易受到数据集中的潜移默化的偏差的影响,这些偏差倾向于积极的结果,从而导致模型对现实的扭曲认知。此外,化学家们通常也不选择冒险的化学反应。为了改进这些AI算法,需要了解什么起作用,以及什么不起作用。这类数据确实存在,但通常只存在于化学家痛苦的记忆中,或是在那些被丢弃的笔记本和尘封的抽屉里,那里存放着无法发表的结果。而去年的一篇计算科学预印本[12]则从理论上证明,在模型训练其他模型的递归循环中,系统的底层真相可能会被“遗忘”,也就是说通过合成数据来人工扩充数据集有可能收效甚微,结果堪忧。Glorius和Grzybowski等人在逆合成AI算法综述中提出的解决方案是更好地利用人类专长。利用我们现有的知识来针对文献中的空白,将比等待计算或现实世界实验提供更多数据来得更快。这与许多开发各种 AI 辅助工作的团队倡导的“将人类置于循环中”的一般原则相呼应。人与人工智能的合作不仅仅是劳动力的分配,而是确保在过程中保留人类的主体性和责任。套用某著名期刊的编者按[15],For chemists, the AI revolutions has
yet to happen,“对于化学家来说,人工智能革命尚未到来”。[1] https://retractionwatch.com/2022/06/09/chemistry-group-at-hokkaido-up-to-three-retractions/
[2] https://cen.acs.org/articles/88/web/2010/11/Bengu-Sezen-Cited-Research-Misconduct.html
[3] https://journals.sagepub.com/doi/full/10.1177/20539517221145372
[4] https://arxiv.org/abs/2307.04340
[5] https://pubs.rsc.org/en/content/articlelanding/2021/sc/d1sc02957f
[6] https://www.annualreviews.org/content/journals/10.1146/annurev-physchem-061020-053438
[7] https://iopscience.iop.org/article/10.1088/1361-648X/abea1c#cmabea1cs6
[8] https://pubs.acs.org/doi/10.1021/acs.jmedchem.1c01984
[9] https://www.science.org/doi/10.1126/science.abj6511
[10] https://www.nature.com/articles/s41586-023-06735-9
[11] https://pubs.acs.org/doi/epdf/10.1021/jacs.4c00338
[12] https://arxiv.org/abs/2305.17493
[13] https://www.chemistryworld.com/features/why-are-computational-chemists-making-up-their-data/4019491.article
[14] https://www.chemistryworld.com/opinion/our-hunger-for-data-could-be-an-unhealthy-diet/4019512.article
[15] https://www.nature.com/articles/d41586-023-01612-x